2月26日,由中国生物信息学学会(筹)、广东省科技基础条件平台中心指导,深圳国家基因库、鹏城实验室、深圳华大生命科学研究院主办的2022『猛犸杯』国际组学数据创新大赛决赛在广州顺利举办。该比赛于去年6月启动,经过初赛、复赛的严格评审筛选,13支队伍从400余个参赛团队中脱颖而出进入本次的最后决战。经过激烈角逐决出最终结果:来自清华大学的“广告位招租”团队和来自重庆医科大学附属第二医院的“重医附二院130岁生日快乐”团队分别荣获时空组学细胞数据分析赛道和精准宏基因组丰度计算赛道的一等奖,另外11支来自北京大学、复旦大学、电子科技大学等高校或机构的队伍分别荣获双赛道的二、三等奖和优秀奖。

沈伟,理学博士,重庆医科大学附属第二医院病毒性肝炎研究所副研究员,研究方向包括:(1)微生物基因组、宏基因组分析方法和工具开发;(2)大规模生物序列搜索;(3)难培养病原微生物检测。主持国家自然科学基金青年项目1项,中国博士后科学基金面上项目1项;以第一作者或共同第一作者身份在Bioinformatics、mSystems、Journal of Genetics and Genomics等期刊发表SCI论文7篇,其中2篇为ESI高被引论文,单篇最高引用962次。
近日,相关研究也以Original paper形式在国际计算生物学会(ISCB)的官方期刊Bioinformatics(影响因子6.931)上发表。该研究开发了针对临床感染样品高通量测序(mNGS)分析和宏基因组分析的原创生物信息学分析算法与软件。
研究论文摘要:
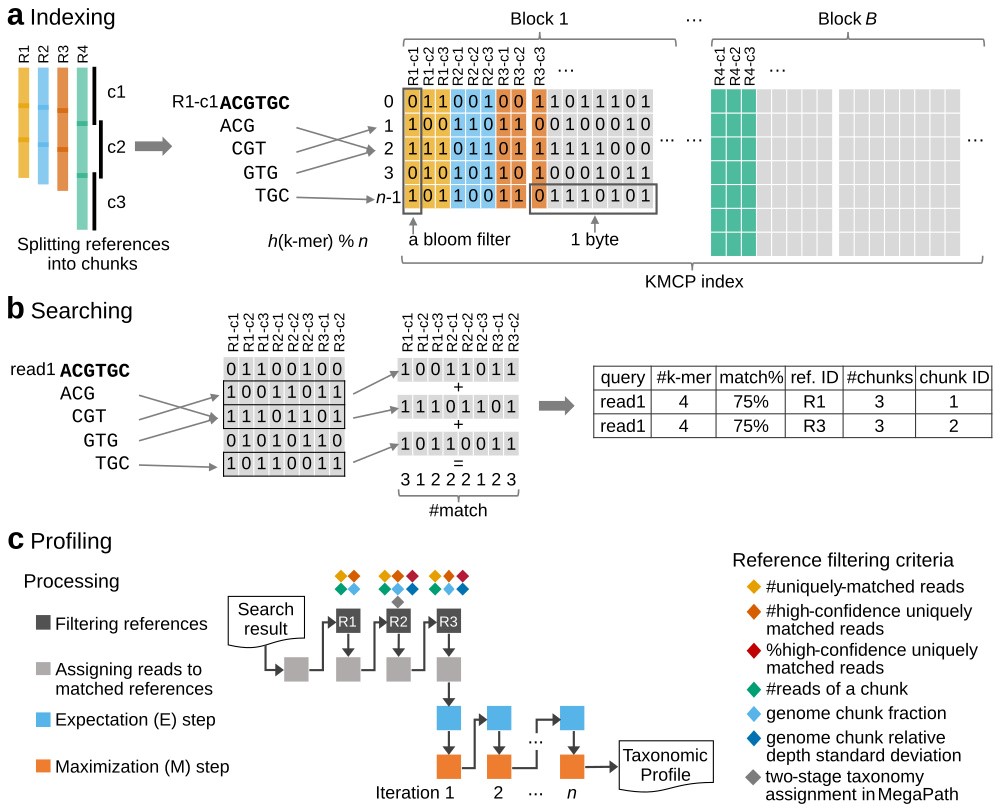
日益增加的微生物参考基因组促进了宏基因组物种组成分析准确性的提高,同时也对宏基因组物种组成分析软件的建库效率、数据库大小、分析时间带来了严峻的挑战。此外,大多数软件主要关注细菌、古菌和真菌,而缺少对病毒群落的关注。本研究中,我们推出KMCP,一个新的基于k-mer的宏基因组物种组成分析软件。它将参考基因组分割成多个相同大小的区块,并将每个区块的k-mer存储在修改和改进的COBS索引中用于序列的快速搜索。KMCP通过结合k-mer相似性和基因组覆盖信息来减少基于k-mer的宏基因组分类和物种组成分析软件的高假阳性。基于模拟和真实数据的基准测试显示,KMCP虽然比其它软件运行时间更长,但是KMCP不仅能在原核生物和病毒群落上准确地分析物种组成,还能在低深度临床样本中提供高可信度的病原微生物检测结果。
论文链接: https://academic.oup.com/bioinformatics/article/39/1/btac845/6965021
